
![]()
Metodología
Muestra
La
población estudiada está conformada por 51 cepas de Enterobacterias
productoras de BLEE, recolectadas entre los años 2002-2004. La
distribución de las mismas fue la siguiente : K. pneumoniae
(25), E. coli (16) , P. mirabilis (2),
Enterobacter spp. (1) , E. cloacae (3), C. diversus
(1), C. freundii (1), Salmonella spp. (1)
y S. marcescens (1). Fueron aisladas de diversas muestras y provenientes
de diferentes servicios de hospitalización, de los siguientes
centros hospitalarios del área Metropolitana de Caracas: Hospital
¨José Gregorio Hernández¨ (23), Policlínica
Metropolitana (6), Maternidad ¨Concepción Palacios¨ (5),
Hospital Militar ¨Dr. Carlos Arvelo¨ (2), Instituto Clínico
¨La Floresta¨ (2), Hospital de Clínicas Caracas (12),
Hospital de Niños ¨J. M. de los Ríos” (1).
Ensayos
de susceptibilidad antimicrobiana y detección fenotípica
de β-lactamasas de Espectro Expandido (BLEE)
Se
llevo a cabo la metodología de Kirby-Bauer recomendada por la
NCCLS 2004. Se determinó el perfil de resistencia a los siguientes
antibióticos: amikacina (Ak), gentamicina (Ge), ciprofloxacina
(Cip), trimetropin/sulfametoxazol (Sxt), piperacilina/tazobactam (Tzp),
amoxicilina/clavulánico (Amc), cefoxitin (Fox), aztreonam (Atm),
ceftazidime (Caz), cefotaxime (Ctx), cefepime (Fep), meropenem (Mem),
imipenem (Imp).
La detección fenotípica de BLEE, se realizó mediante el método de doble difusión en disco10 y por el recomendado por la NCCLS 2004.
Los controles de calidad utilizados fueron: E. coli ATCC 25922-ATCC 35218 y Pseudomonas aeruginosa ATCC 27853, Klebsiella pneumoniae ATCC-700603.
Detección
molecular de BLEE mediante ensayos de Reacción en Cadena de la
Polimerasa (PCR)
Para
realizar el PCR para detección de BLEE se trabajó según
el esquema de Chanawong y col. (2000), utilizando células bacterianas
completas. Los iniciadores utilizados para la detección de BLEE
son mostrados en la Tabla 1.
Iniciador No. |
Nombre |
Secuencia (5´- 3´) |
1 |
SHV |
TCAGCGAAAAACTTG 24 |
2 |
SHV |
TCCCGCAGATAAATCACCA 24 |
3 |
TEM:Mab F |
GGGGAGCTCATAAAATTCTTGAAGAC 13 |
4 |
TEM:Mab R |
GGGGGATCCTTACCAATGCTTAATCA 13 |
5 |
CTX-M-2F |
ATGATGACTCAGAGCATTCG 3 |
6 |
CTX-M-2R |
TTATTGCATCAGAAACCGTG 3 |
Tabla
1 Secuencia de Iniciadores para Tipificación de BLEE |
El protocolo utilizado para la reacción de PCR fue el siguiente: 4 min a 95 ºC, 30 ciclos de 1 min a 94ºC (desnaturalización), 1 min y 30 seg a 55ºC (alineamiento), 1 min a 72ºC (polimerización), y una extensión final de 10 min a 75ºC. Los reactivos y volúmenes utilizados están enumerados en la Tabla 2.
Reactivos |
Volúmenes |
Buffer PCR 10X |
2,5 μl |
MgCl 2 25 mM |
2 μl |
dNTPs 10 mM* |
1 μl |
Iniciador 1 100 mM |
1 μl |
Iniciador 2 100 mM |
1 μl |
Mezcla de H 2 O/Taq Polimerasa** |
16,5 μl |
Suspensión celular |
1 μl |
Volumen final |
25 μl |
Tabla
2 Reactivos y volúmenes utilizados en la reacción de PCR * Solución 10 mM para adenina, timina, citosina y guanina ** Mezcla: 0,2 μl de Taq Polimerasa y 16,3 μl de agua |
Los productos de amplificación fueron visualizados mediante electroforesis en gel de agarosa al 1,2%. Se utilizó el marcador de PM ladder 100 pb. El control positivo para BLEE tipo SHV: K. pneumoniae M-1740 . Para BLEE tipo TEM: E. coli M-3195 . Para BLEE tipo CTX-M: K. pneumoniae M-1803. El control negativo: E. coli ATCC 25922. Todas las cepas controles positivos fueron donadas por el Dr. Marcelo Galas del Instituto de Enfermedades Infecciosas-ANLIS. Buenos Aires-Argentina.
La caracterización de BLEE tipo SHV se llevó a cabo mediante ensayos de PCR-RFLP (reacción en cadena de la polimerasa y análisis de polimorfismos mediante el uso de enzimas de restricción).
Una vez obtenido el producto de amplificación del gen codificante para ß-lactamasas tipo SHV, se procedió a cortar el ADN con la enzima de restricción Nhe I. Esta enzima corta una secuencia especifica de nucleótidos generada en la posición 238 del gen, producto del cambio mutacional G----A, esta mutación es característica de varias BLEE derivadas de SHV-1 (SHV-2, SHV-5, SHV-12). De esta manera se logró determinar que el producto de amplificación correspondía a una BLEE derivada de SHV-1.
Transferencia
plasmídica de genes que codifican para BLEE y de Integrones clase
I
La
transferencia de plásmidos que codifican para BLEE y que portan
genes estructurales correspondientes a Integrones clase I, se llevó
a cabo mediante ensayos de Conjugación Bacteriana en medio sólido.
Las cepas donantes corresponden a un numero de 36 cepas de Enterobacterias
que poseían los marcadores fenotípicos adecuados para
dicho ensayo, es decir, presentaban sensibilidad a Rifampicina y/o Ácido
nalidíxico. La cepa receptora de plásmidos conjugativos
fue la Escherichia coli K-12 NCTC50170, RC-711 rif (donada
por el CVCM). La selección de cepas transconjugantes fue realizada
en placas de medio agar Luria-Bertani (agar LB) suplementadas con rifampicina
(40 µg/mL) y ampicilina (30 µg/mL) y una segunda placa de agar LB suplementadas
con rifampicina (40 µg/mL) y ceftazidime ó cefotaxime (10 µg/mL).
Aislamiento
plasmídico
Para
el aislamiento de plásmidos se utilizó el método
de lisis alcalina, empleando polietilenglicol y cloruro de litio 4 .
En el caso de cepas transconjugantes se utilizó la técnica
a mediana escala y para las cepas donantes a pequeña escala,
ambos cultivos con 24 horas de crecimiento en agitación a 37°C.
El aislamiento plasmídico fue visualizado mediante electroforesis en gel de agarosa al 0,6%. Se utilizó el marcador de peso molecular del bacteriófago Lambda Hind III.
Detección
de Integrones clase I mediante ensayos de Reacción en Cadena
de la Polimerasa (PCR)
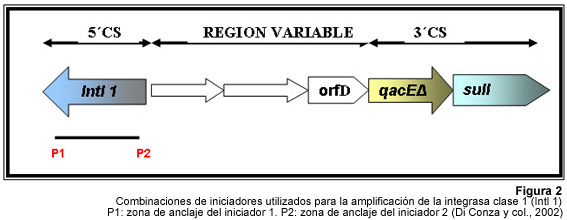
Se
utilizaron iniciadores (Tabla 3) que reconocen secuencias específicas
de la integrasa clase 1 (Figura 2), la detección de la integrasa
se llevó a cabo a partir de las células bacterianas de
las Enterobacterias y sus correspondientes transconjugantes y del ADN
plasmídico aislado, según el protocolo de Di Conza y col.
(2002)7 . Los volúmenes de reactivos fueron similares
a los usados en el PCR de identificación de BLEE.
El protocolo utilizado para la reacción de PCR fue el siguiente: 6 min a 94 ºC, 30 ciclos de 45 seg a 95ºC (desnaturalización), 45 seg a 55ºC (alineamiento), un minuto a 72ºC (polimerización), y una extensión final de 10 min a 72ºC.
Iniciador No |
Nombre |
Secuencia (5--> 3) |
Posición en la secuencia |
No. De Acceso |
1* |
IntI1-15 |
ACC GCC ACC TTT CAG CAC AT 7 |
1339-1358 |
M95287 |
2* |
IntI1-13 |
GCG TTC GGT CAA GGT TCT GG 7 |
2266-2247 |
M95287 |
Tabla
3 |
El control positivo utilizado fue Lisado celular obtenido de la cepa HUC-0117, la cual presenta un integrón clase 1 14 y el negativo fue un lisado celular preparado a partir de la cepas de Escherichia coli K-12 ó Escherichia coli ATCC 25922, las cuales no presenta ningún integrón.
![]()